Tárgyak
Összegzés
A rosszul vizsgált pikornavírus, a humán parechovírus 3 (HPeV3) újszülöttek szepszisét okozza rendelkezésre álló terápiák nélkül. A HPeV3 4,3 Å felbontási struktúrája önmagában és 15 Å felbontással humán monoklonális antitest Fab komplexekben mutatja be a picornavírus kapszid várható szerkezetét, három különálló tulajdonsággal. Először is, a HPeV3 RNS genomjának 60% -a 60 helyen erősen rendezett, amit az aszimmetrikus rekonstrukció megerősít, és kölcsönhatásba lép a VP1 és VP3 kapszid fehérjék konzervált régióival. Másodszor, a VP0 N kifejezés stabilizálja a kapszid belső felületét, ellentétben más pikornavírusokkal, amelyeknél az ejekciónál, mint a VP4, RNS transzlokációs csatornát képez. Végül a VP1 hidrofób zsebe, az antipicornavirális gyógyszer, a plekonaril kötőhelye blokkolva van, és így alkalmatlan az antivirális fejlődésre. Ezek az eredmények együttesen irányt mutatnak a semlegesítő antitestek, az RNS-fehérje kölcsönhatásokon alapuló vírusellenes gyógyszerek és az RNS nukleáción alapuló vírus-összeállítás boncolásához.
Bevezetés
A Picornaviridae egy kicsi, pozitív érzékű, ikozaéderes szimuláció, egyszálú RNS vírusok családja. A parechovírus az a faj, amely 16 genotípussal rendelkezik ezen a családon belül, és főleg az emberek, különösen a gyermekek enyhe fertőzésével társul. Azonban egy kialakuló kórokozó, a humán parechovírus 3 (HPeV3) súlyos fertőzéseket okozhat a központi idegrendszerben, például az agyhártyagyulladást 1, és az újszülöttek szepszisének 2 vezető oka. A HPeV-fertőzés leküzdésére nincsenek vírusellenes szerek vagy oltóanyagok. Sok más pikornavírustól eltérően a HPeV-k mind struktúrájuk, mind funkciójuk szempontjából gyengén jellemezhetők, a HPeV1 kivételével, ahol a 3 receptor ismert. A HPeV3 által a HPeV3 által mutatott nagy különbségek a tropizmusban a többi HPeV-hez képest elengedhetetlenné teszik a HPeV3 szerkezeti tulajdonságainak vizsgálatát annak patogenezisének és lehetséges receptorkötésének jobb megértése érdekében.
Krioelektron mikroszkóppal és képrekonstrukcióval elemezzük a HPeV3 szerkezetét önmagában és humán Fab monoklonális antitesttel komplexben. A virion szerkezete azt mutatja, hogy a VP1 zsebkötő gyógyszerek, mint pl. A pleconaryl, valószínűleg nem kötődnek a HPeV-hez; hogy a VP0 fontos fehérje a kapszid belső felületének stabilizálásához, és végül, hogy a HPeV-összeállítást valószínűleg a genom és a kapszid többszöri interakciója vezérli, a VP1-ben és a VP3-ban konzervált aminosavak, valamint az RNS-ben lévő szár- és hurokszerkezetek révén. Elkülönítjük és jellemezzük a HPeV3-ra specifikus humán monoklonális antitestet, amely nagyon hasznos lehet a vírusok diagnosztizálásának elősegítésében és a vírus-gazda kölcsönhatások tanulmányozásában.
Eredmények és vita
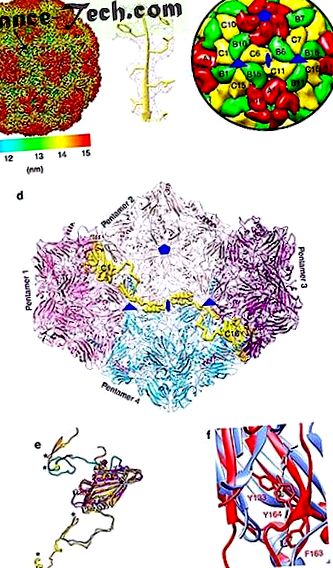
A HPeV3 szerkezete
Teljes méretű kép
A HPeV3 RNS rendezett szerkezete
Teljes méretű kép
Teljes méretű kép
Hidrofób VP1 zseb
A vírusellenes gyógyszer, a plekonaril és származékai általában blokkolják az enterovírus fertőzéseket, például az EV-D68 (4. hivatkozás). Ezek a gyógyszerek kiűzik a VP1 β-hordó hidrofób zsebében található lipidet ("zsebfaktor"), és a 4-es kapszid stabilizálásával blokkolják az RNS-bevonatot. A HPeV3 VP1 hidrofób zsebébe vezető csatornát három nagy konzervált Y133, F163 és Y164 oldallánc blokkolja (1f és 3. ábra) az EV-D68-hoz képest (4. hivatkozás). Ezért megmagyarázhatjuk azokat a megfigyeléseket, amelyek szerint a pleconaril nem működik a 2. klinikán, és megjósolhatjuk, hogy az ilyen zsebfaktor-utánzatok nem fognak működni az egyik eddig szekvenált HPeV ellen.
HPeV3 - Fab komplex szerkezet
Elkülönítettünk egy HPeV3-specifikus humán monoklonális antitestet, az AT12-015-öt, amely az 152037, A308/99 HPeV3 izolátumokhoz és két fertőzött sejtben lévő klinikai izolátumhoz kötött. Nem semlegesítette az 152037-et egy Vero sejtvonalon alapuló vizsgálatban. Az 152037 HPeV3 izolátum szerkezetét a Fab AT12-015 komplexben oldottuk meg (1. kiegészítő táblázat; 4a. Ábra; 1. kiegészítő ábra). A Fab-molekulák felismernek egy konformációs epitópot a kanyon peremén (4a. Ábra). A hozzájárulások mind a VP3-ból (hZβB, βChA, βEβF és βGβH hurkok), mind pedig a VP1-ből (βBβC, βCβD és βEβF, C) származnak, főként a HPeV3-ban konzerválva, ami megmagyarázza az antitest specifitását erre a genotípusra (3. ábra). . A Fab ujjlenyomat átíveli a VP1 C terminust, ahol sok más HPeV tartalmaz RGD motívumot az integrin receptoraikhoz való kötődéshez (3. és 4b. Ábra) 3. Mivel az AT12-015 specifikus a HPeV3-ra és nem kötődött a többi vizsgált HPeV-genotípushoz, nagyon hasznos lehet a vírusdiagnosztika elősegítésében és a vírus-gazda kölcsönhatások tanulmányozásában. A semlegesítő páciensszérumok epitópjaival történő további összehasonlítás segíthet megérteni a betegek semlegesítési mechanizmusát 13 .
( nak nek ) A HPeV3-Fab AT12-015 komplex sugárirányú színfelületi ábrázolása 15 Å felbontás mellett, 1,5σ-val az átlagos küszöb felett. A Fab-molekulák (vörös) a kanyon régió körül kötődnek. ( b ) HPeV3 ütemterv. A Fab lábnyom (piros körvonal) a VP3-ra (zöld) és a VP1-re (piros) illeszkedik.
Teljes méretű kép
Összességében ez a munka több stabilizáló RNS-kapszid kölcsönhatást mutat a parechovírus gyógyszerek új célpontjaként. Feltárjuk ezen kapszulák szokatlan stabilitását intra-pentamer szinten VP3 kölcsönhatások és az inter-pentamer VP0 kölcsönhatások révén, és javasoljuk, hogy a HPeV genom bevonó mechanizmusa jelentősen eltérjen a többi pikornavírustól.
Mód
Vírustisztítás és inaktiválás.
A HPeV1-Harrist és a HPeV2-2008-at a Holland Nemzeti Egészségügyi és Környezetvédelmi Intézet, Bilthoven, Hollandia biztosította. A HPeV4-251176, a HPeV5-552322, a HPeV6-550389 és két HPeV3 klinikai izolátumot az Amszterdami Akadémiai Orvosi Központ Klinikai Virológiai Laboratóriumából kaptuk. A vírusállományokhoz a HPeV1, HPeV2, HPeV3 A308/99 (Hiroyuki Shimizu és Miyabe Ito ajándék típusa) és a HPeV4-6 sejteket HT29 sejtvonalban tenyésztettük Eagle minimális esszenciális táptalajában, L-glutaminsavval (0, 2 ×), nem esszenciális aminosavak (1x), sztreptomicin (0,1 μg ml -1), penicillin (0,1 μg ml -1) és 2% hő-inaktivált FBS (a HT29 sejteket 8% FBS hővel inaktivált táptalajban tartottuk). Ezeket a törzseket alkalmaztuk az AT12-015 antitest kötési és semlegesítési vizsgálatokban.
Antitestek előállítása és Fab fragmensek előállítása.
Az 152037 HPeV3 izolátum elleni AT12-015 humán monoklonális antitestet emberi vérből állítottuk elő. Röviden: az emberi memória CD27 + IgG + B sejtjeit AIMSelect módszerrel tenyésztettük olyan egészséges donoroktól, akik egy évvel korábban megerősített, izolált 152037-es HPeV3 fertőzésből gyógyultak fel. Az egyes sejteket szubklónoztuk olyan B-sejtkultúrákból, amelyekben a felülúszó immunfluoreszcenciával mutatott kötődést a HPeV3-fertőzött sejtekhez. RNS-t izoláltunk monoklonális B-sejtekből az antitest nehéz és könnyű lánc szekvenciájának kinyerése céljából. Egyedülálló szekvenciákat alkalmaztunk rekombináns fehérje előállítására 293 T-sejtben, majd az IgG1 antitesteket tovább tisztítottuk HiTrap protein A oszlopokkal, ÄKTA (GE) eszközön. Az AT12-015 Fab fragmenseket Pierce Fab mikromop készlet segítségével állítottuk elő a gyártó utasításai szerint az antitest teljes emésztésének elérése érdekében. A kapott Fab-fragmenseket HPeV3 kapszidákkal 60: 1 mólarányban összekevertük, és 30 percig inkubáltuk 37 ° C-on, 1x TNM-ben, hogy a vírus-Fab komplex képződjön, mielőtt üvegesítettük volna.
HPeV semlegesítési vizsgálat
A HPeV3 152037 izolátum AT12-015-vel történt semlegesítését úgy vizsgáltuk, hogy Vero-sejteket előre inkubált vírusokkal (100 TCID50 egység) fertőztünk meg változó mennyiségű antitesttel (0,03–7,5 µg ml -1) 37 ° C-on 1 órán át. A fertőzött sejteket 24 óránként 7 napig monitoroztuk a CPE megjelenése szempontjából. A 7. napon RNS-t extraháltunk a felülúszóból egy teljes nukleinsav-izoláló készlet alkalmazásával, a MagnaPure LC készülékkel (Roche Diagnostics), a reverz transzkripciót és a cDNS-t (komplementer DNS) használtuk a vírus kópiaszámának valós idejű PCR-vel történő becsléséhez. LC480 műszer (Roche Diagnostics) 16. Az AT12-015 képességét az 152037 HPeV3 izolátum semlegesítésére BGM (bivaly zöld majom vese), A549 (humán vastagbél adenokarcinóma) és Caco2 (humán vastagbél adenokarcinóma) sejtrétegekben is tesztelték. Ezenkívül az AT12-015 által végzett HPeV1, HPeV2, HPeV4, HPeV5 és HPeV6 elleni kereszt-semlegesítést Vero sejtekben is tesztelték. A HPeV1-6 fertőzött sejtek immunfluoreszcenciáját az AT12-015 alkalmazásával is igazoltuk.
Krio-elektron mikroszkópia és képfeldolgozás.
A formaldehiddel inaktivált, tisztított HPeV3-at üvegesítettük úgy, hogy a minta 3 µl-ét a Quantifoil R3.5/1 rácsokra vittük fel, 2 másodpercig szárítottuk 92% relatív páratartalom mellett, és Leica EM GP segítségével folyékony etánba merítettük. A rácsokat FEI Titan Krios transzmissziós elektronmikroszkóppal vizsgáltuk, Cs-re korrigálva 300 keV-on. A képeket Falcon II detektoron rögzítették alacsony dózisú körülmények között, 59 000 × névleges nagyítással, 1,14 Å/pixel mintamérettel. Képenként hét képkockát gyűjtöttünk számlálási módban az FEI EPU automatizált egy részecske-gyűjtő szoftverrel. A filmkereteket az utómunka előtt a motioncorr 17 szoftver segítségével kezdetben egymáshoz igazították.
Az aszimmetrikus rekonstrukció érdekében a legjobb 41 845 részecske orientációját további 16 fordulóra finomították a Relion háromdimenziós automatikus finomítási opciójával, szimmetria megkötése nélkül. Két független adatsor közötti végső finomítás 10,36 Å felbontást eredményezett az FSC 0,143 kritérium alapján.
A HPeV3-Fab AT12-015 komplexek alikvot részeit hőkezelt, rendkívül finom szénrétegen, réz- és szénrácsokon (TED PELLA) üvegesítettük, saját készítésű giljotin alkalmazásával. A rácsokat FEI Tecnai F20 transzmissziós elektronmikroszkóppal vizsgáltuk 200 keV feszültségen, Gatan 626. kriosztát alkalmazásával. A képeket Kodak SO-163 filmeken rögzítettük alacsony dózisú körülmények között, 50 000 × névleges nagyítással. A filmeket Zeiss szkennerrel (Photoscan) digitalizáltuk 7 µm lépésmérettel, 1,4 pixel pixelméretként. Minden mikrográf kontrasztátviteli függvényét a fent leírtak szerint becsültük meg. A részecskéket négyszeres időközönként mikrográfiákon gyűjtöttük össze, a RobEM 21 program segítségével, 101 dobozos dobozmérettel. 150 részecskéből véletlenszerű indítási modellt állítottunk elő az AUTO3DEM v 4.05.1 (21. hivatkozás) alkalmazásával. A teljes adatsort kettéválasztották, és az egyes halmazok által generált független rekonstrukciókból meghatározták a tájolást és az eredetet. Az 564 részecskéből 15 Å végső felbontását a 0,143 FSC kritérium alapján értük el.
A modell felépítése
A három HPeV3 kapszid fehérje szerkezetét több sablon összehasonlító modellezésével jósoltuk meg az I-TASSER 25 szerver segítségével. A VP0 sablonstruktúrái tartalmaztak egy felhasználó által definiált sablont, amely a VP4 humán enterovírus (PDB ID: 3vbf) N terminál egyesített szerkezetét tartalmazta az emberi enterovírus VP2 (PDB ID: 3vbo) 26 öblítésére, valamint a száj- és körömfájást. vírus (PDB ID: 1qqp) 27, poliovírus 1 (PDB ID: 1pov) 28, szarvasmarha enterovírus (PDB ID: 1bev) 29, 1 echovírus (PDB ID: 1ev1) (30. hivatkozás) és hepatitis A (PDB ID: 4qpg) ) 31. A legjobb modell C pontszáma –1,03 volt.
A VP3-hoz egy üres emberi enterovírus felhasználó által biztosított sablont (PDB ID: 3vbo) 26 használtak, a program által kiválasztott következő sablonok mellett: coxsackievirus A9 (PDB ID: 1d4m) 32, echovirus 1 (PDB ID: 1d4m) PDB: 1ev1) (30. hivatkozás), humán enterovírus (PDB ID: 3vbf) 26, humán enterovírus (PDB ID: 3vbh) 26, humán coxsackievirus A16 (PDB ID: 4jgy) 7. A legjobb modell C pontszáma –0,61 volt.
A VP1 esetében üres emberi enterovírust (PDB ID: 3vbo) 26 adtak külső sablonként a program által kiválasztott következő sablonok mellett: humán enterovírus 71 (PDB azonosító: 3vbh) 26, humán enterovírus 71 (PDB azonosító: 3zfe) 33, humán coxsackievirus A16 (PDB ID: 4jgy) 7, emberi enterovírus 71 (PDB ID: 4cdq) 34, coxsackievirus A 9 (PDB ID: 1d4m) 32, humán rhinovírus 14 (PDB ID: 1ncq) 35. A legjobb modell C pontszáma –0,75 volt.
A homológiai modelleket szigorúan illesztették a HPeV3 térképre az UCSF-Chimera 36 "fit to map" funkciójával. Az UCSF-Chimera 36 'zónázási' funkciójának felhasználásával a HPeV3 kapszid térképet aszimmetrikus egységre osztották 6 Å sugarú merev illesztésű VP0-VP3-VP1 modell segítségével. A VP0-VP3-VP1 modell rugalmasan illeszkedett az aszimmetrikus egységbe két iMODfit 37 és FlexEM 38 flex illesztési program eredményeinek összehasonlításával, hogy konszenzusos illeszkedést érjünk el 39. Mindkét rugalmas beállító programot az alapértelmezett beállításokkal használták. Ezután a mintákat manuálisan finomítottuk a Coot 40-ben, a csonkolással, ahol a sűrűség nem volt látható, a gerinc illeszkedésének javításával és a fenilalanin, tirozin, triptofán és arginin maradványok nehéz oldalláncainak beállításával, ahol látható volt. A modelleket tovább finomították a Phenix 41 valós terének finomításával. Az összes képi megjelenítést az UCSF-Chimera 36-ban végeztük .
Az RNS strukturált régió egyetlen példányát a Bulk Eraser alkalmazásával extraháltuk az UCSF-Chimera 36-ban. A kivont térfogat körülbelül 30 nukleotidot tartalmazhat. Az izolált HPeV3 152037 genom (GenBank ID: GQ183026) 3 'transzlálatlan végén lévő 30 nukleotid régiót (7181-7210) hajtogattuk az RNAfold 42 webszerverre, és a hajtást az RNAComposer 43-ban modelleztük. Ezt az RNS modellt illesztették a kivont kötethez az UCSF-Chimera 36 'Fit to Map' opciójával .
Az AT12-015 lábnyomot úgy becsültük meg, hogy a HPeV3 atommodellt ráhelyeztük a HPeV3-Fab AT12-015 komplex rekonstrukciójára, és elkészítettük az atommodell útitervét a mesés lábnyommal, mint a RIVEM 44 kontúrvonalaként. Az összes aminosavmaradékot, amelyet a felületen kitettek e lábnyomon belül, a Fab epitópjainak tekintettük.
további információ
Belépési kódok: A Az ikozaedrikusan szimmetrikus HPeV3, az aszimmetrikus HPeV3 és a HPeV3-Fab AT12-015 sűrűségtérképeit az Elektronmikroszkópos Adatbankban helyezték el EMD-3137, EMD-3322 és EMD-3138 hozzáférési kódokkal. A HPeV3-hoz igazított modelleket az európai fehérjebankban helyezték el a PDB ID: 5APM alatt. A HPeV3 rekonstrukcióhoz összegyűjtött összes nyers adat elérhető az EMPIAR-10033 hozzáférési kódú elektronmikroszkópos kísérleti képfájlon keresztül.
Hogyan idézhetem ezt a cikket: Shakeel, S. és mtsai. Az újszülött szepszist okozó kialakulóban lévő pikornavírus nagy felbontású struktúrájában több kapszid stabilizációs kölcsönhatás mutatkozott meg. Nat. Comun. 7: 11387 doi: 10.1038/ncomms11387 (2016).
- Papaya, egy gyümölcs, amelynek számos egészségügyi előnye van, és amelyet a napi étrendbe be kell építenie -
- A sperma többszörös és hasznos tulajdonságai Placeres S Moda EL PAÍS
- Mi az ideális kalóriabevitel egy 5 méter magas, 95 kg súlyú ember számára?
- A fogyni vágyó nőknek magas tejfehérje-tartalmú étrendet kell fogyasztaniuk -
- Kilenc gyümölcs, számos előnnyel a bőr számára